plasmid DNAの物性
2本鎖DNAを輪っか状にデザインした物をPlasmid DNA (pDNA)と言います。完全なpDNAでは、輪ゴムをよじった状態(super coil)となり輪っか状ではなくなります。そのことで、見かけ上の分子量は小さくなります。もしも、2本の内1本に切断箇所(ニック)が存在すると、super coilではなくなり、輪っか状になります。さらに、もう一方の鎖に切断箇所があると、輪っか状も崩れて、直鎖のDNAの形態になります。完全体であるsuper coilのpDNAは、以上の状態の変化による性質の違いを利用して精製されます。
ベクター・デザイン
既存のベクターをデザインするには、既存の知られた遺伝子部品を組み合わせることが基本です。リンクした以下のサイトは、標準のプラスミド・ベクターや、AAVベクター、レンチウイルス・ベクターなど、数十種類のベクターをバックボーンにして、好みのベクターをデザインすることができます。
ベクター・ビルダー
https://www.vectorbuilder.jp/design.html
plasmid DNAの抽出方法
plasmid DNA (pDNA)の製造で最もクリティカルな工程は、その抽出です。pDNAを生産する細胞にE.coliを用いた場合、E.coliのgenomeと目的のpDNAを効率よく分離抽出することが重要です。pDNAは、スーパーコイル(輪ゴムが更によじれている物をイメージするとわかりやすい)となっており、E.coliのgenomeとは物理的強度が異なることを利用して、アルカリ抽出やガラスピーズのミル抽出が一般的に行われます。
- pDNAには耐性があると言って、アルカリ抽出でもガラスビーズ・ミル抽出でも分解されない訳ではないため、pDNAを効率よく抽出するためには、その処理条件の最適化が必要となる
- pDNAは負電荷であるため、その精製は、AEXが基本となるが 12)、疎水クロマト(HIC)も適応できる。
- silica単体にも塩基性アミノ酸バッファで結合させることが可能である
- 工業的な生産では、沈殿化工程は、遠心機を使用するよりフィルター処理する方法が好まれる。フィルター工程の最適化も重要な検討項目である
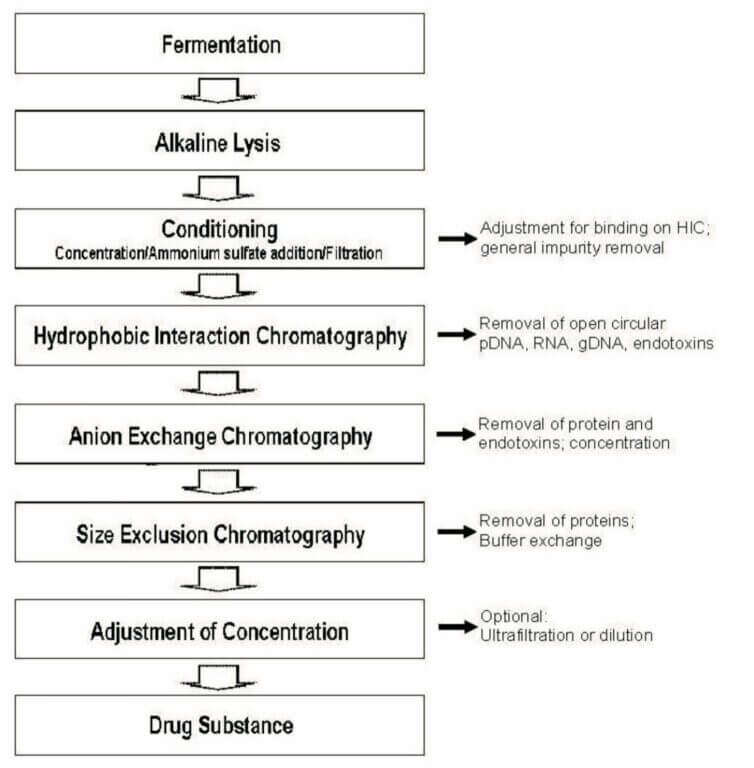
生産フロー
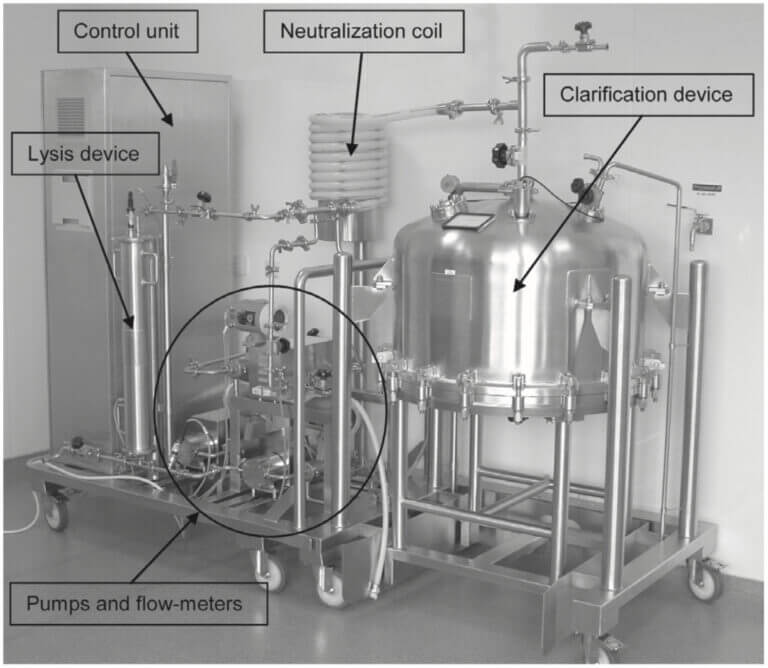
ベーリンガー社が、CDMOとしてpDNAの効率的な製造方法を考案している。参考文献 3)、その主たる内容は、1~200L規模のcGMPに適用できるpDNAの製造において、高い効率で抽出できるアルカリ抽出およびビーズを用いた方法である。
- Vectorのデザイン
- copy数
- plasmidサイズ (不要な配列の除去)、可能な限り小さく
- 耐性遺伝子から栄養要求遺伝子への変更
- Cell Bankの作成
- 拡大培養
- 生産培養
- ハーベスト
- アルカリ溶解 (Alkaline Lysis)
- pH12
- ガラスビーズ in Tubeによる破砕(マイルドに!)
- supercoiled plasmidは、機械的ストレスに弱い
- 中和(上清にplasmid、沈殿にタンパク質やゲノムDNA)
- pH12
- ろ過システムによる濾過
- 清澄システムにもガラスビースが充填されている
- クロマトグラフィー
- HIC
- Capturing step for pDNA
- binding and elution condition
- removing of endotoxin RNA, genome DNA

- AEX
- binding for pDNA
- removing of entoxine
- SEC
- recovery : 1mg/mL
- HIC
- UF/DF
- concentration : 10mg/mL, but higher viscocity
- Bulk Fill
plasmidの培養方法
参考文献 1)より。大腸菌を使って生産させるのが一般的です。工業的には生産性が高いためです。
| Item | pVGXI1, pVGXI2 |
| Size | 4.2kb, 4.6kb |
| bad case | OD < 15, 0.6g plasmid/10L fermentor |
| optimization | OD >40, 2.4 g ~ 3.0 g plasmid/10L fermentor |
| 考慮事項 | pVGXI2は、高いpoly A, 低いGC比率 |
その他の精製方法として、以下のフローも参考の一つとして示します。特記する点は、ろ過助剤により細胞を効率的に回収している点である。
- 培養条件 : 詳細は参考文献1)を参照
- 流下培養、生産性 0.24g/L
- ハーベストには、ろ過膜、ろ過助剤(珪藻土+ミネラル;ベントナイト)を使用。
- 抽出・溶解工程
- pDNAの選択的沈殿化
- 再溶解/ろ過
参考文献 1)
1) Large Scale Production and Scale Up of DNA Plasmid Vectors With Complex Gene Inserts (2014)
プラスミドベクターを大腸菌で大規模に生産するために、(1)細胞株の最適化と (2)配列の最適化を実施した。
プラスミドpVGXI1(4.2kb)は、最初は10L発酵スケールで増殖は不良(OD <15、プラスミド収量:0.6g / 10L)であった。 細胞株の最適化により改善することができたOD> 40、プラスミド収量2.4g / 10L)。 このプロセスは、100Lおよび400Lスケールまで効果的にスケールアップ可能であった。
プラスミド、pVGXI2(4.6kb)は、その配列の高いA残基の繰り返しと低いGC%で構成されていたが、 3つの異なる大腸菌細胞株で10Lスケールで検討した結果、各細胞株では、増殖は不良(OD <10)であり、プラスミドがほとんど生成されなかった(プラスミド収量:0.7g / 10L)。
配列の最適化を行うことにより、pVGXI2は、標準的な大腸菌株で高い増殖性(OD> 50)およびプラスミド収量(3g / 10L)を10Lスケールで確認できた。
さらに、プロセス最適化により、これらの2つのプラスミドは、発酵収率が4倍に改善し、スケールアップも達成できた。さらに、これらのプラスミドの精製により、臨床使用に適した高品質のDNA産物を得ることができた。
https://www.cell.com/molecular-therapy-family/molecular-therapy/fulltext/S1525-0016(16)35244-3
参考文献 2)
2) 医薬品グレードの大規模プラスミドDNA製造プロセス(2015)
プラスミドDNAの製薬用途には、直接的または間接的に、特定の品質基準が必要です。ヒトの「適正製造基準」(GMP)グレードへの直接遺伝子導入は必須ですが、ウイルスベクター(AAVなど)などのGMP生産では、使用するプラスミドDNAを必ずしもGMPで生産する必要はないとの記載があります。このような規制の側面に加えて、研究室規模(最大数ミリグラム)から工業規模(ミリグラムからグラム規模)へのプラスミドDNA生産プロセスの拡大がここで扱われる問題です。
https://pubmed.ncbi.nlm.nih.gov/24715291/
参考文献 3)
3) プラスミドDNAの工業生産 (2008)
ベーリンガーの新しいcGMP生産システムに関する。pDNAの抽出方法は、アルカリ抽出法とビーズミル抽出法の併用であると理解しました。本当のところ、詳細については不明なのでわかりません。
プラスミドDNAを製造する場合、適正製造基準は慎重なベクター設計から始まります(図1)。真核生物のプロモーター、遺伝子配列、およびポリA部位は主に治療効果に影響を与えますが、ベクターの残りの部分は製造にとって重要です。
ベクターバックボーンのすべての要素、機能、および特性は、プロセスの堅牢性と製品の品質に関して評価する必要があります。
ベーリンガーインゲルハイムオーストリアは、天然のColE1 / pUCoriに基づいた抗生物質を含まないプラスミド選択のためのホストベクターシステムを開発しました。
生産性は、2.4g/L
supercoiled plasmidについての記載あり。培養終了時には、90% supercoiled plasmidであるpDNA均一性を目標とする。
https://www.genengnews.com/magazine/86/industrial-manufacturing-of-plasmid-dna/
pDNAはスーバーコイル
参考文献 4)
4) Efficient Disruption of Escherichia coli for Plasmid DNA Recovery in a Bead Mill (2017)
概要 – 要約:ビーズミルによるpDNAの抽出に関する研究。総pDNA(pDNA(t))およびスーパーコイル状pDNA(pDNA(sc))について、抽出に関するモデルを開発を目的に、ミル頻度、セル濃度、およびビーズサイズの2つのレベル23要因を検討。
その結果は、応答曲面法によって分析した。
pDNA(t)の最適化抽出条件として、13.26 mg / g dcw(93.41%の回収率)、30 Hzのミル周波数、0.10〜0.25 mmのビーズサイズ、および20 g wcw / Lのセル濃度を決定。
pDNA(sc)の最適化抽出条件として、7.65 mg / g dcw(92.05%の回収率)、15 Hzのミル周波数、0.10〜0.25 mmのビーズサイズ、10 g wcw / Lのセル濃度を決定。
考察 – ビーズミルでの細胞破壊は、アルカリ処理と比較して、pDNA(t)およびpDNA(sc)の放出に効率的であることが証明された。
https://res.mdpi.com/d_attachment/applsci/applsci-08-00030/article_deploy/applsci-08-00030.pdf
アルカリ抽出
参考文献 5)
5) Hot-Alkaline DNA Extraction Method for Deep-Subseafloor Archaeal Communities (2014)
アルカリ処理と加熱を使用した新しいDNA抽出方法をの検討。 1 M NaOHで98°Cで20分間処理すると、さまざまな深さで収集された海底下の堆積物サンプル中の微生物細胞の98%以上が破壊されました。 しかし、DNAの完全性試験では、このような強アルカリ性および熱処理により、DNA分子がPCRでは増幅できない短い断片に切断されることが示されました。
その後、アルカリ条件と温度条件を最適化して、DNAの断片化を最小限に抑え、高い細胞破壊効率を維持しました。 最良の条件は、さまざまな深さからの海底下の堆積物サンプルで50〜80%の細胞破壊率を生み出し、完全な16S rRNA遺伝子(すなわち、約1,500 bp)の増幅に十分なDNAの完全性を保持しました。 最適化された方法では、従来のキットベースのアプローチを使用した抽出と比較して、テストしたすべてのサンプルでより高いDNA濃度も得られました。 リアルタイムPCRと細菌および古細菌の16SrRNA遺伝子のパイロシーケンスを使用した比較分子分析は、新しい方法が古細菌DNAとその多様性の増加をもたらしたことを示し、従来の方法よりも海底下の微生物群集のより良い分析範囲を提供することを示唆しています。
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3957647/
参考文献 6)
6) A continuous process to extract plasmid DNA based o Alkaline Lysis (2007)
ここで紹介するプロトコルは、アルカリ溶解プロトコルに基づくスケーラブルな連続プロセスでプラスミドDNAを抽出します。このプロセスでは、採取した細菌を2つの混合チャンバーに制御された速度で通過させて、溶解を行い、アルカリ度を制御します。得られた溶液は、一連の フィルターで汚染物質を除去し、エタノールで沈殿させます。 このプロセスは、粗プラスミドを取得する前にすべての遠心分離ステップを置き換え、より多くの量の需要を満たすために簡単にスケールアップできます。 この手順を使用して、プラスミドを抽出し、50分から90分で、4リットルの大腸菌培養物から精製することができます。 プラスミドの収量は80〜90mg/L 培養です。
細胞の形質転換→細胞増殖→プラスミドの抽出と精製。最も一般的な抽出方法はアルカリ抽出。連続プロセスに適用するには、pDNAの大規模な調製のためのアルカリ溶解法のスケールアップです。 細胞破壊の代替方法は沸騰溶解です。 しかし、このアプローチでは、pDNA収量と純度に一貫性がなく、メソッドも複雑です。最近、熱溶解プロトコルに基づく連続プロセスの開発と最適化の成功を報告しましたが、溶解後も遠心分離ステップが必要でした。アルカリ溶解手順によるpDNAの大規模抽出は、特に次の理由により、問題があると認識されています。 プロセスで使用される3つの溶液、それぞれ0.4M NaOH/C2コンテナ、2% SDS/C3コンテナ、3 M potassium acetate, 5 M acetic acid, and pH 4.8/C4コントなに地蔵しておく。Bacteria懸濁液(100 OD/TE buffer)とC2+C3のミックス液を混和してアルカリ溶解させ、その後、C3の溶液と混和することで、不純タンパク質とGenomeを沈殿化させる。濾液を回収し、70%EtOHで沈殿化させて濾過膜で沈殿を回収する。
Flow rate : 3cm/sec
遠心分離ステップを排除するために、0.2 μmの中空糸カートリッジを使用して細胞を回収。1)細胞溶解後の破片を除去し、プラスミドがエタノールで沈殿した後のRNA汚染を除去するための70μmナイロンフィルター。中空糸カートリッジはさまざまなサイズで製造できるため、適切なカートリッジを選択して、必要な最終容量を回収できます。 発酵細菌。 溶液IIIと混合した後、溶解したバクテリアを孔径が徐々に小さくなる4層のナイロンフィルター(375、186、122、70 μm)に通して、このサイズ範囲の破片やその他の不純物を除去します. 70μmのナイロンフィルターは、エタノール沈殿後のプラスミドとRNAの分離に最も効果的。
スペルミジン圧縮18、CTAB沈殿19、20、ゲルろ過12、磁気ビーズ精製21などのさまざまな後続の下流精製プロトコルに容易に採用できます。 このプロセスは、従来のアルカリ溶解の問題を回避し、DNAワクチン接種、非ウイルスベクターベースの遺伝子治療、RNAi発現コンストラクトおよびその他の関連する臨床および研究アプリケーションのニーズを満たすプラスミドDNAの自動生成のためのプラットフォームを提供します。 この連続システムは操作が簡単ですが、システムのセットアップと最適化には、接続、チューブサイズ、同期ポンプの調整を含む最初のステップが必要です。 流速、混合セルのサイズ、および振とう頻度は、新しい実験条件に合わせて最適化する必要があります。 発酵プロセスとバクテリアの収穫は、収穫されたバクテリアのアルカリ溶解から始まる以下の手順では説明されていません。 発酵は参考文献に記載されているように実施する必要があります。 15、および細菌は、参考文献に記載されているように、このプロトコルで使用するために収穫する必要があります。 15、TEの代わりにソリューションIを使用
https://www.researchgate.net/publication/5576598_A_continuous_process_to_extract_plasmid_DNA_based_on_alkaline_lysis
プラスミド抽出の原理
参考文献 7)
7) いまさら聞けないプラスミド抽出法の原理 (2011) – 生物工学第89巻 –
アルカリ変性の進み具合は、分子量の大きな染色体と、plasmid DNA(pDNA, スーパーコイルになっているものは特に安定的)とでは異なる。アルカリ抽出の原理を知ることができる。
1. covalently closed circular : ccc (物理的に最も安定)
2. open circular : oc
3. liner : lブラスミドベクターによってコピー数が1桁も違う
https://www.sbj.or.jp/wp-content/uploads/file/sbj/8909/8909_yomoyama_1.pdf
1. pUC, pBluescript, pGEM, pTZ, pBR322, pACYC, pSC101
プラスミド抽出の特許
参考文献 8)
8) US Patent for Plasmid DNA extraction process Patent – Justia Patents – プラスミドDNA抽出プロセス – FujifilmDiosynth Biotechnolgies UK Limited特許 (2010/07/29出願、US特許8889852)
フロースルー装置を用いて、95℃~120℃、10秒未満、5秒を超えて120℃超えないこと。pDNAは通常3kbp~4kbpの範囲。pH4~pH10,好ましくはpH7~9(0~100mM Tris-HCl)。カルシウムなどの細胞壁のカチオンを可溶化するEDATなどのキレート剤を含み得る。ボリオール(スクロースなど)によるpDNAの放出促進として、5%~10%を含み得る。界面活性剤X-100を1~10%を含み得る。カオトロープとして尿素を0.5~8M、好ましくは1~3M。細胞溶解剤リゾチームなどを必要としないが、必要に応じて使用し得る。
https://www.sbj.or.jp/wp-content/uploads/file/sbj/8909/8909_yomoyama_1.pdf
What is DNA?
参考文献 9)
9) What is DNA? – QIAGEN –
ウイルス、細菌のgenomeの長さ(bp)と分子量。
pDNAのAEX、Silica-membraneおよびマグネットSilicaによる精製。
https://www.qiagen.com/jp/service-and-support/learning-hub/molecular-biology-methods/dna/
プラスミド抽出
参考文献 10)
10) Plasmid vs. Genomic DNA Extraction: The Difference (2014)
ゲノムDNA抽出 (gDNA)は、最大限の抽出方法で行います。まず、酵母、植物、細菌の場合、溶解には、原形質膜を機械的に破壊する前に、強くて硬い細胞壁を酵素的に破壊することが含まれます。細胞壁は通常、細胞壁ペプチドグリカンを加水分解するリゾチームとセリンプロテアーゼプロテイナーゼKで消化されます。特定のグラム陽性種の場合、リゾスタフィンは酵素消化をさらに促進します。細胞壁の組成が異なる外来種には、異なる酵素を使用する必要がある場合があります。
その後、機械的な細胞壁破壊は、gDNA抽出のためのより普遍的な溶解方法として、ビーズビートがあります。0.1mmのガラスビーズまたは0.15mmの細かいガーネットビーズを使用して、ミル装置で行います。丈夫な糸状菌(例えば、アスペルギルスおよびフザリウム属)の場合、細胞材料は液体窒素で急速冷凍され、乳棒と乳鉢で粉砕された後、適切な溶解バッファーを含む溶液中で急速にボルテックスされます。
精製は、シリカへの結合を促進するグアニジン塩を添加したフェノール-クロロホルムまたはスピンフィルター膜技術を使用して行うことができます。
大腸菌の染色体は、細胞当たり約0.005ピコグラムにのぼる、サイズはわずか4.5メガバイトです。単一の開始コロニーからの典型的な一晩培養物は、約1-2× 109細胞/ mlを含みます。理論的には、これは、1mlの培養物が109個の細菌細胞あたり約5µgのgDNAを生成することを意味します。選択したアプリケーションに必要なDNAの量を計算するときは、これを考慮に入れてください。
プラスミドDNA(pDNA)はgDNAから分離しておく必要があるため、プラスミドDNAの抽出は少し注意が必要です。この分離はサイズに基づいており、適切な分離は適切な溶解方法の使用に依存します。
pDNA抽出の場合、細胞壁を酵素でかみ砕いたり、ガラスビーズで叩いたりするよりも、溶解をはるかに微妙にする必要があります。BirnboimとDolyは、1979年にアルカリ溶解によるプラスミドDNA抽出のための(事実上)普遍的な方法を発明しました。
溶解バッファーには水酸化ナトリウムとSDSが含まれており、プラスミドとgDNAを完全に変性させます(つまり、DNAを一本鎖に分離します)。過度の変性は不可逆的にプラスミドを変性させる可能性があるため、このステップを迅速に実行することが重要です。次に、サンプルを酢酸カリウム溶液で中和してプラスミドを再生します。これは、プラスミドとgDNAを分離するための鍵です。プラスミドは小さいため、簡単に再アニーリングしてdsDNAを形成できます。ただし、ゲノムDNAは長すぎて完全に再アニーリングできず、代わりに絡み合って相補鎖が分離されたままになる傾向があります。遠心分離中、gDNA(タンパク質に結合)はペレットを形成しますが、プラスミドDNAは可溶性のままです。このステップでは、gDNAが壊れやすいため、サンプルを激しくボルテックスしたり混合したりしないことが重要です。壊れたgDNAは、再アニーリングしてプラスミドと溶解するのに十分小さい場合があります。
pDNAの精製、次に、上清中のプラスミドDNAをエタノール沈殿するか、フェノール-クロロホルムまたはスピンフィルターを使用して精製します。スピンフィルターテクノロジーを使用している場合、中和バッファーにはグアニジン塩が含まれているため、ライセートはシリカに直接結合してさらに洗浄および溶出できます。得られたDNAは、ほとんどの下流の分子生物学アプリケーションにとって十分に純粋です。
トランスフェクションにプラスミドが必要な場合は、陰イオン交換精製が汚染エンドトキシンを除去するためのより良い選択です。より高速なシリカベースの精製セットアップを使用して、エンドトキシンの除去も可能です。高収量のプラスミドDNAを単離するには、対数増殖期後期または定常期初期の培養物を使用します。プラスミド維持のために適切な濃度の新鮮な単一コロニーと新鮮な選択抗生物質を使用して培養物を準備します。プラスミド抽出物にgDNAが混入する可能性があるため、細菌培養物を増殖させないことが重要です。
https://bitesizebio.com/1660/plasmid-v-genomic-dna-extractionthe-difference/
プラスミドのシリカゲルによる精製
参考文献 11)
11) DNA Adsorption to and Elution from Silica Surfaces: Influence of Amino Acid Buffers (2014)
正に帯電した(アルギニン(ARG)とヒスチジン(HIS))、負に帯電した(アスパラギン酸(ASP)とグルタミン酸(GLU))、極性中性(GLU)に分類されるアミノ酸を含む溶液からシリカへのDNA吸着への影響を測定しました。アスパラギン(ASN)、グルタミン(GLN)、セリン(SER))、および非極性疎水性(ロイシン(LEU)、プロリン(PRO)、およびグリシン(GLY))。さらに、DNA抽出の固相として2種類のシリカ粒子を選択しました。最初のタイプであるMagPrepシリカコーティング磁性粒子(Merck)は、核酸の固相抽出に一般的に使用されます。ただし、これらの粒子のシリカ表面は、核酸抽出用に最適化されている可能がある
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4097040/
プラスミド精製特許
参考文献 12)
12) プラスミド精製 JP2007500711A – 特許 –
多孔のポアサイズを持つ陰イオン交換レジンによる精製。大きいサイズのプラスミドは、外表面に結合、プラスミドより小さいサイズの負電荷物は、プラスミドがアクセスできないボア内に結合させる。
背景情報には、>80%飽和硫酸アンモニウムによりpDNAを可溶性、ゲノムDNAを不溶性に分離できる特許情報の記載がある(米国特許第6214586号(Genzyme Corp.)
https://patents.google.com/patent/JP2007525222A/ja
pDNAのCTAB精製
参考文献 13)
13) Milligram scale parallel purification of plasmid DNA using anion-exchange membrane capsules and
a multi-channel peristaltic pump (2007)低濃度CTAB (0.1-4g/L) 沈殿法によるpDNAとRNA/Endotoxinの分離 : 2g/L or 10g/L CTAB/50mM NaCl溶液を添加し、Incubation20分, cfg(38,000xg 20min, 20℃)/pptを70% EtOHで洗浄し、0.6M NaCl, 25mM Tris-HCl, 1mM EDTA, pH7.4で氷冷下で溶解。
https://www.researchgate.net/profile/Stefan-Schmidt-27/publication/6232344_Milligram_scale_parallel_purification_of_plasmid_DNA_using_anion-exchange_membrane_capsules_and_a_multi-channel_peristaltic_pump/links/59de01f545851557bde325bd/Milligram-scale-parallel-purification-of-plasmid-DNA-using-anion-exchange-membrane-capsules-and-a-multi-channel-peristaltic-pump.pdf
pDNAのHIC精製
参考文献 14)
14) Purification of plasmid (pVaxLacZ) by hydrophobic interaction chromatography (2005)
2.5M AmSO4(硫酸アンモニウム)で沈殿化した後、HICでBindingさせ、1.5M AmSO4でElutionする。
https://www.scielo.br/scielo.php?script=sci_arttext&pid=S1516-89132005000400014
参考文献 15)
Using a single hydrophobic-interaction chromatography to purify pharmaceutical-grade supercoiled plasmid DNA from other isoforms (2012)
1. HICカラムにより抽出したpDNAを不純物であるタンパク質、RNA、エンドトキシンなどの宿主汚染物質から分離精製する
2. scpDNAとocpDNAを分離可能。
3. 3 M硫酸アンモニウムの存在下では、scpDNAはocpDNAよりも疎水性が高くなり、ocpDNAは最初にHICから溶出できることで、一部のocpDNAをscpDNAから除去可能
4. 効果的な免疫応答と感染性チャレンジからの保護を引き出すために高いスーパーコイルレベルが必要であることを示されている(Cupillard et al。、2005; Pillai et al。、2008)
5. 製品には細菌ゲノムDNA、RNA、タンパク質、エンドトキシンが含まれていない必要
6. 第二に、pDNA産物は高い均質性でなければなりません。つまり、ほとんどのプラスミドはスーパーコイルアイソフォーム
7. プラスミドの品質分析(アガロースゲル電気泳動、HPLCなど)は、最終pDNAが98±1.2%のスーパーコイル率と検出限外以下の不純物であった。
8. リポソームトランスフェクション実験は、ここで説明する精製pDNAがコントロールpDNAよりも高いトランスフェクション効率を持っていることを示しました。 したがって、この研究で提示された方法は、医薬品グレードのpDNAを製造するための主な要件を満たしている。 ラボ規模の準備から大規模な生産への移行は簡単です。
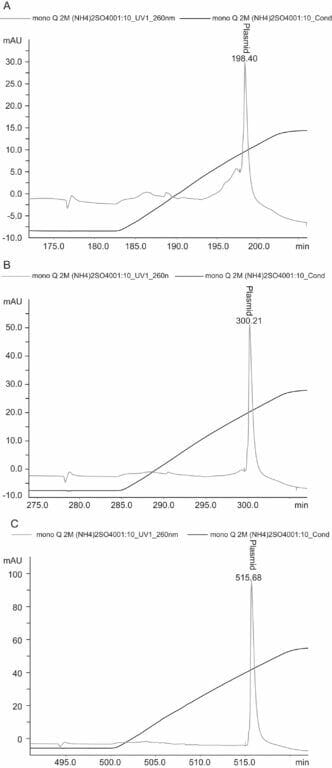
Figure 3. Plasmid purity analysis by HPLC. Plasmid purity was determined by anion-exchange HPLC on a MONO QTM 5/50 GL column. A linear gradient was performed at 0.5 mL/min by increasing the NaCl 0–1 M in 40 mM Tris/HCl, pH 8.0 for 10 CV at a flow rate of 0.5 mL/min. (A) plasmid sample purified by 2 M ammonium sulfate, (B) plasmid sample purified by 2.5 M ammonium sulfate, (C) plasmid sample purified by 3 M ammonium sulfate.
https://www.tandfonline.com/doi/full/10.3109/13880209.2012.703678
参考文献 16)
Improved downstream process for the production of plasmid DNA for gene therapy (2005)
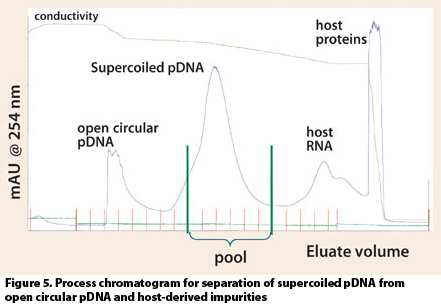
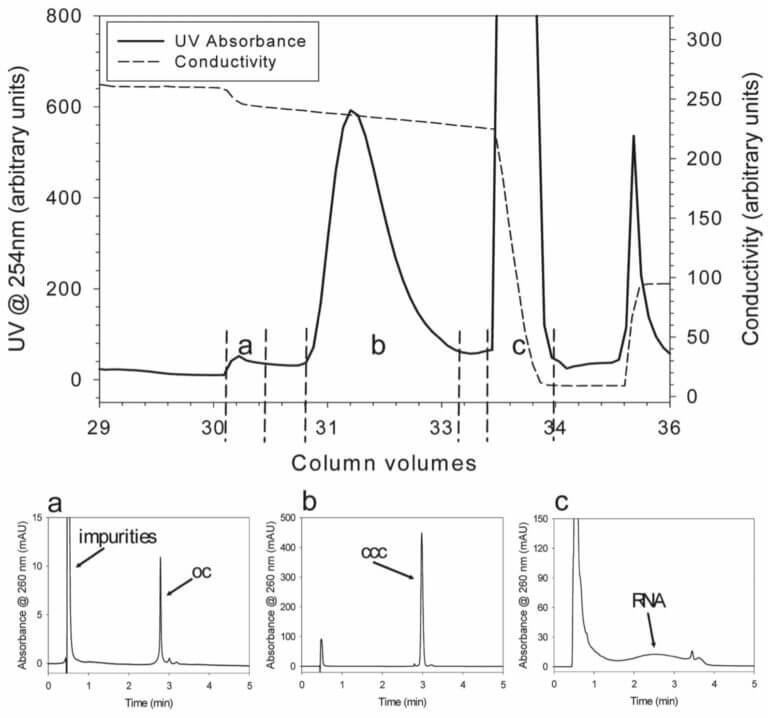
HICにより、OCは最初のピークに、RNAは後のピークに、スーパーコイルであるpDNAは、その間に溶出される。
Figure 4. Performance of the HIC step.
Top: Elution profile of the HIC step (pRZ-hMCP1, 4.9 kbp). Main fractions are indicated by dashed lines. Bottom: Analyti- cal HPLC chromatograms of the HIC fractions; a) the first HIC peak contains impurities and open circular (oc) pDNA; b) the second HIC peak contains mainly supercoiled (ccc) pDNA; c) residual RNA and further im- purities elute when conductivity is suddenly strongly decreased. More strongly bound material is washed out of the column dur- ing regeneration (peak at the end of the HIC chromatogram
分析は、AEC。
http://www.actabp.pl/pdf/3_2005/703.pdf
プラスミドバックボーン
参考文献 17)
プラスミドバックボーン – Plasmid backbones –
日本語訳:
https://parts.igem.org/Plasmid_backbones
プラスミドバックボーン・キット
参考文献 18)
蛍光レポーターまたはカスタム配列をターゲット部位に挿入するために用いるドナープラスミド構築キットEdit-R HDR Plasmid Donor Kit – FUNAKOSHI –
https://www.funakoshi.co.jp/contents/63964
核酸デリバリー
参考文献 19)
遺伝子治療のための新規人工遺伝子デリバリーシステム : 多機能性エンベロープ型ナノ構造体の開発 (2007) – YAKUGAKU ZASSHI 127(10) 1685-1691 (2007) –
https://yakushi.pharm.or.jp/FULL_TEXT/127_10/pdf/1685.pdf
参考文献 20)
[GS02-1] 脾臓選択的遺伝子導入キャリアの開発とDNAワクチンへの応用 – 日本薬学会 第141年会
免疫細胞へ効率的に抗原遺伝子を送達し、発現させることが課題となる。脾臓は多くの免疫細胞 が局在し、全身性免疫を司る臓器であり、DNAワクチンの有望な標的臓器と考えられる。我々が見出した LNPは、脾臓選択的な遺伝子発現を示し、脾臓内において抗原提示細胞に分布していた。
発表表紙はパスワード必要
編集履歴 2020/10/22 Mr.HARIKIRI 2020/10/25 追記 (内容の充実化) 2020/11/19 追記 (ベクター・デザイン) 2020/12/24、追記 (pDNAの物性) 2021/01/05 誤字脱字訂正 2021/05/10 追記 (文献19,20)