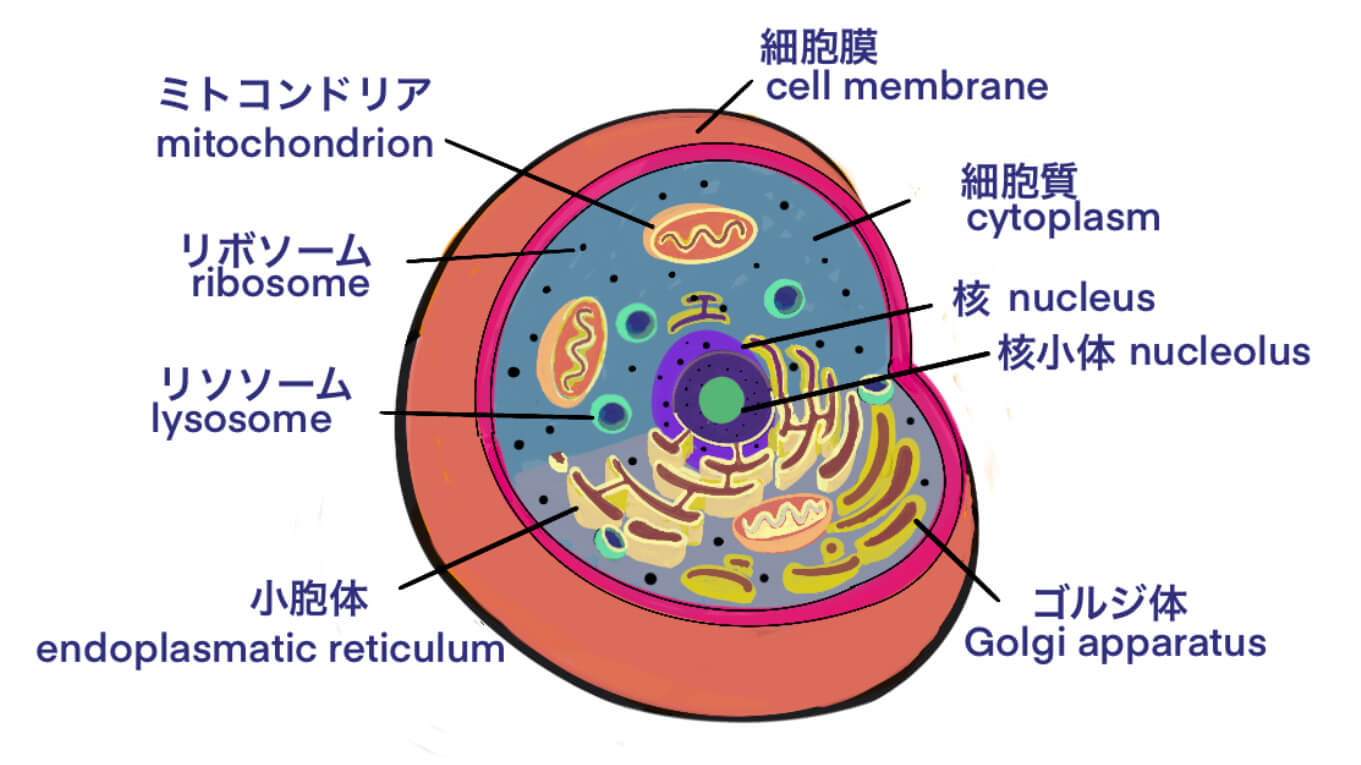
タグ: education
-
[Bio-Culture] 大腸菌; E.coliの増殖率は細胞長の増大に関わる [2021/02/27]
Post Views: 1,056 大腸菌の増殖率 一般的に、大腸菌の増殖率(growth rate)、1時間…
投稿者

-
[Bio-Culture] Poloxamer 188 (Pluronic F-68) – バイオリアクターの撹拌翼による細胞のダメージを抑制 [2021/02/24]
Post Views: 1,151 Poloxamer 188 Poloxamer 188という名称は、馴染み…
投稿者

-
[Bio-Edu] イオン交換クロマトグラフィの原理 – バイオロジクスの製造における基本 [2021/02/13]
Post Views: 1,051 イオン交換の原理を使ったクロマトグラフィ 文字通りイオンを交換反応で結合で…
投稿者

-
[Bio-Edu] タンパク質の「イオン交換体」による精製原理 – 目的タンパク質の物性を知る必要性 [2021/02/25]
Post Views: 794 はじめに タンパク質の精製とは、どのような作業をするのでしょうか? 精製とは目…
投稿者

-
[用語] pDNA ; Plasmid DNA – 染色体とは異なる遺伝子/環状/自己複製/F Plasmid/R Plasmid/ Vector Plasmid [2023/03/25]
Post Views: 1,038 plasmid DNAとは pDNA ;Plasmid DNA, 輪っか状…
投稿者

-
[Bio-Edu] ウイルスの等電点(pI) – 組換えAAVでは,empty/fullの違いでpIは異なる – 分離精製は工業化の課題である[2021/02/23]
Post Views: 1,226 はじめに 遺伝子治療用のウイルス・ベクターとしてAAVを使用した場合、目的…
投稿者

-
[Bio-Edu] DNAとRNAの違い – レジメ – [2020/10/11]
Post Views: 1,051 ID24191 DNAとRNAの違い DNAはRNAの設計図になります。R…
投稿者

-
[Bio-Education] 医薬品の開発スケジュール [2021/04/08]
Post Views: 990 医薬品の開発スケジュールの概要 バイオロジクスの開発スケジュールは、以下のよう…
投稿者

-
[Bio-Education] 細胞のモノクローナル化のための基本原理 – 最新装置を中心に解説 – [2020/09/03]
Post Views: 960 ID22024 細胞のモノクローナル化のための3つの基本操作 細胞をモノクロー…
投稿者